Цифровое пространственное профилирование GeoMx DSP — анализ транскриптомов
Понимание неоднородности тканей имеет решающее значение для ответа на ключевые биологические вопросы в трансляционных исследованиях. Нынешняя парадигма анализа тканей требует компромисса между морфологическим и молекулярным высокомультиплексным анализом, жертвуя ценной информацией или потребляя ценные образцы. В медицинской практике все больше уделяется внимания проектам, связанным с генетикой и онкологией, так как это открывает новые возможности для диагностики и лечения различных заболеваний. Качество каждой медицинской услуги в наше время зависит от использования современных научных подходов и инструментов в биологии и генетике. Цифровое пространственное профилирование GeoMx DSP – уникальная разработка 2019 года не имеющая аналогов, которая позволяет проводить количественный анализ экспрессии множества генов (до полного транскриптома) и белков на стекле в выбранном участке.
Для полного транскриптома генов путем секвенирования важным этапом в получении информации является подготовка библиотек на основе исходного материала – РНК после удаления рибосомальной РНК или обогащения поли-А+. Эти библиотеки специально разработаны для таких задач, как подготовка стандартных РНК-библиотек, покрывающих всю длину мРНК для проведения sense mRNA-seq. В состав наборов для получения библиотек входят все необходимые компоненты, которые гарантируют точность и эффективность секвенирования. Технология замещения цепи stop/ligation используется при создании библиотек с помощью этих наборов, обеспечивая высокое качество и надежность полученных данных. Таким образом, анализ полного транскриптома с использованием этих библиотек позволяет получить результат с информацией о всей генетической активности в исследуемой системе.
Выбор области интереса можно проводить различным образом: геометрически – обвести область интереса фигурой, сегментно – выбрать только области генов, в которых связалась та или иная флуоресцентная метка, контурно – отметив слои или контуры вокруг различных участков, сеточно – настроив параметры сетки или просто отметив единичные клетки.
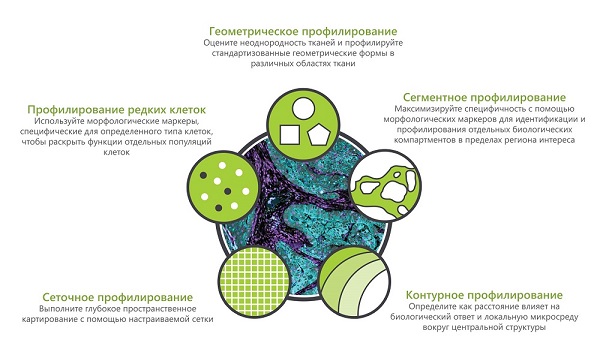
Основные преимущества технологии исследования единичных клеток:
- Высокая мультиплексность – количество до 800 мишеней при работе в комбинации c системой nCounter, до полного транскриптома генов при работе с системами высокопроизводительного секвенирования (NGS)
- Минимальное количество образца - один срез для анализа РНК или белков в гене
- 4-ех цветная визуализация для определения морфологического контента
- Возможен повторный анализ слайдов при работе с белками
- Настраиваемая разрешающая способность – выбор участка интереса от 600 микрон до единичной клетки
- Высокая пропускная способность – до 20 срезов в день
- Количественный подсчет мишеней
Адаптация технологии NanoString для количественного анализа белков
В результате растущего интереса к гетерогенности опухолей и редких клеточных подгрупп (например, циркулирующих опухолевых клеток) высокочувствительный метод, позволяющий профилировать несколько клеток или даже отдельные единичные клетки с высоким уровнем мультиплексирования, вызывает все больший интерес. Штрих-кодирование гена ДНК было разработано и применено к растворимым белкам [2-7]. Тем не менее, применение технологии к единичным клеткам или клеточным суспензиям было ограничено из-за сложностей обнаружения и амплификации штрих-кода в клеточной микросреде. Эти ограничения были первоначально превзойдены развитием свето-опосредованного клеточного штрих-кодирования (LCMB). Agasti и соавторы [8] разработали метод протеомного профилирования на основе антител, который позволял обнаруживать белки в суспензиях живых клеток с помощью антител, конъюгированных с уникальными олигомерами ДНК, через линкер, расщепляемый под воздействием ультрафиолета. Эксперименты продемонстрировали линейную корреляцию олигонуклеотидов, отделившихся от антитела против HER2, к количественной оценке клеток SK-BR3, 3T3 и MDA-MB-231 на основе проточной цитофлуориметрии. Было показано обнаружение отдельных клеток SK-BR3 в экспериментах с разведением и обнаружение EGFR, EpCAM и HER2 / neu, которые можно получить при мультиплексном окрашивании линий клеток рака молочной железы.
В то время LMCB был значительным прогрессом, метод был полуколичественным, и амплификация с помощью ПЦР освобожденных олигонуклеотидов ограничивала мультиплексирование до 5 мишеней и приводила к сдвигам. Метод ПЦР для анализа освобожденных олигонуклеотидов был впоследствии заменен на метод NanoString [9]. Детектирование олигонуклеотидов системой nCounter устранило необходимость проведения амплификации и увеличило количество одновременно анализируемых мишеней. Эксперименты по белковому профилированию продемонстрировали обнаружение до 90 белков в свежих и замороженных биопсийных образцах. Дальнейшая валидация показала сравнимые результаты с окрашиванием коньюгированными и неконьоюгированными антителами, воспроизводимось, идентичность окрашивания против индивидуальных маркеров и окрашиванием коктейлем против 60 аналитов , высокую корреляцию с уровнем экспрессии 6 маркеров (CD44, HER2, EGFR, CA19-9, CK7, MUC1) полученных методом проточной цитофлуориметрии. Метод конъюгации антител был впоследствии адаптирован компанией NanoString, которая разработала специализированный прибор для визуализации образцов ткани и отбора проб с выбранных пользователем участков для последующего анализа на приборах nCounter и SPRINT – Система цифрового пространственного профилирования GeoMx Digital Spatial Profiler.
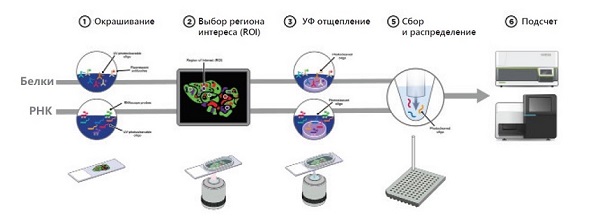
Весь протокол состоит из 6 шагов (описан протокол при анализе экспрессии белков):
- Окраска слайда антителами, конъюгированными с олигонуклеотидами через линкер, расщепляемый под воздействием ультрафиолета
- Выбор интересующих областей интереса (Regions of Interests – ROI): геометрически выбранная область, фенотипически выбранная область, единичная/единичные клетки
- Облучение заданной области и отщепление олигонуклеотидов
- Захват олигонуклеотидов с помощью микрокапилляра и распределение в планшет
- Подсчет и анализ экспрессии
Система цифрового пространственного профилирования может работать как со свежими тканями, так и зафиксированными тканями, заключенными в парафин (FFPE)
Химическая и техническая сложность платформы GeoMx предполагает необходимость надежной проверки ее данных: технических характеристик, чувствительности и воспроизводимости. Исследования показали, что технология имеет больший динамичесмких диапазон чем ИГХ, обладает высокой воспроизводимостью и хорошо коррелирует с методами «золотой стандарт». Более подробно обо всей технической валидации и возможностях системы в исследовании «New tools for pathology: a user’s review of a highly multiplexed method for in situ analysis of protein and RNA expression in tissue» Jérémie Decalf , Matthew L. Albert , James Ziai [1]
Система позволяет анализировать большое число маркеров при небольшом количестве материала. Так в исследованиях 2018 года сотрудниками Netherlands Cancer Institute производилась оценка экспрессии белков биопсийного материала меланомы для понимания иммунного статуса и оценке потенциального ответа на терапию. Данные показали, что инфильтрация иммунными клетками коррелирует с ответом на терапию [10] Группа из MD Anderson Cancer Center также занимались поиском маркеров при меланоме, которые бы показали ответ на терапию ниволумабом (nivolumab) или комбинации ниволумаба (nivolumab) и ипилимимаба (ipilimumab). Технология позволила группе количественно измерить экспрессию белков в частности в ин фильтрующих опухоль иммунных клетках до и вовремя лечения в образцах тонкоигольной биопсии. Благодаря технологии было выявлено, что наличие популяций иммунных клеток и их активационный статус могут прогнозировать клинический ответ на терапию [11]. С целью определения механизмов взаимодействия между генетическими структурами, ученые изучают профиль генома в разных условиях жизни человека. В ходе изучения генома ученые обнаруживают различные механизмы связи между генетическими структурами, что помогает определить наиболее эффективные методы исследования.
На данный момент доступны для заказа:
1) Готовые белковые панели:
| Панель Human Immuno-Oncology | Панель Mouse Immuno-Oncology | Панель Human Neuroscience |
|---|---|---|
| Rb IgG | Rb IgG | Tdp-43 |
| Ms IgG1 | Rt IgG2a | APOE |
| Ms IgG2a | Rt IgG2b | Tau |
| PD-1 | PD-1 | Phospho-Tau (S404) |
| Histone H3 | Histone H3 | P2RX7 |
| CD68 | MHC II | Amyloid-Beta 1-40 |
| HLA-DR | CD11b | Amyloid Precursor Protein |
| S6 | S6 | Phospho-Tdp-43 (S409/S410) |
| Ki-67 | Ki-67 | Amyloid-Beta 1-42 |
| Beta-2-microglobulin | F4/80 | Ubiquitin |
| CD11c | CD11c | Rb IgG |
| CD20 | CD19 | Ms IgG1 |
| CD3 | CD3e | Ms IgG2a |
| CD4 | CD4 | S6 |
| CD45 | CD45 | Histone H3 |
| CD56 | CD8a | CD68 |
| CD8 | CTLA4 | HLA-DR |
| CTLA4 | GZMB | CD11b |
| GZMB | GAPDH | MAP2 |
| GAPDH | PD-L1 | Ki-67 |
| PD-L1 | PanCk | CD163 |
| PanCk | SMA | Synaptophysin |
| SMA | Fibronectin | IBA1 |
| Fibronectin | LAG3 | GFAP |
| CD45RO | OX40L | Myelin basic protein |
| FOXP3 | Tim-3 | NeuN |
| CD34 | VISTA | S100B |
| CD66b | B7-H3 | Olig2 |
| FAP-alpha | GITR | CD40 |
| CD14 | CD127/IL7RA | CD45 |
| CD163 | CD27 | GAPDH |
| 4-1BB | CD40 | Neurofilament light |
| LAG3 | CD40L | CD31 |
| OX40L | ICOS | CD39 |
| Tim-3 | CD44 | P2ry12 |
| VISTA | CD86 | TMEM119 |
| ARG1 | CD14 | Alpha-synuclein |
| B7-H3 | CD163 | Phospho-Alpha-synuclein (S129) |
| IDO1 | CD31 | ApoA-I |
| STING | CD34 | LRRK2 |
| GITR | FOXP3 | Park5 |
| CD127 | Ly6G/Ly6C | Park7 |
| CD25 | BatF3 | Tyrosine Hydroxylase |
| CD80 | CD28 | Calbindin |
| ICOS | IFNGR | PINK1 |
| PD-L2 | Epcam | FUS |
| CD40 | S100B | Phospho-Tau (T231) |
| CD44 | ER | Phospho-Tau (S396) |
| CD27 | AhR | Phospho-Tau (S199) |
| MART1 | AR | ADAM10 |
| NY-ESO-1 | GFP | Phospho-Tau (S214) |
| S100B | Pmel17 | PSEN1 |
| Bcl-2 | Her2 | NRGN |
| EpCAM | Neprilysin | |
| Her2 | IDE | |
| PTEN | BACE1 | |
| ER-alpha | C4B | |
| PR | CD11c | |
| BAD | CD9 | |
| BCL6 | CLEC7A | |
| BCLXL | CSF1R | |
| CD95/Fas | CTSD | |
| GZMA | GPNMB | |
| NF1 | MERTK | |
| Cleaved Caspase 9 | EMP1 | |
| p53 | Vimentin | |
| PARP | ATG12 | |
| BIM | ATG5 | |
| BRAF | BAG3 | |
| EGFR | LC3B | |
| Phospho-c-RAF (S338) | GBA | |
| Phospho-JNK (T183/Y185) | HSC70 | |
| Phospho-MEK1 (S217/S221) | LAMP2A | |
| Phospho-p38 MAPK (T180/Y182) | TFEB | |
| Phospho-p44/42 MAPK ERK1/2 (T202/Y204) | P62 | |
| pan-RAS | VPS35 | |
| p44/42 MAPK ERK1/2 | ||
| Phospho-p90 RSK (T359/S363) | ||
| Phospho-AKT1 (S473) | ||
| Phospho-GSK3B (S9) | ||
| Phospho-GSK3A (S21)/Phospho-GSK3B (S9) | ||
| INPP4B | ||
| PLCG1 | ||
| Phospho-PRAS40 (T246) | ||
| Phospho-Tuberin (T1462) | ||
| Pan-AKT | ||
| MET |
2) РНК панель для работы с системами nCounter
| База Immune Pathways | |||||
|---|---|---|---|---|---|
| 41-BB | CD40 | EPCAM | IL15 | STAT1 | VISTA |
| AKT1 | CD40LG | FAS | ITGAV | STAT2 | OAZ1 |
| ARG1 | CD44 | FOXP3 | ITGB2 | STAT3 | POLR2A |
| B2M | CD45 | GZMB | ITGB8 | TBX21 | RAB7A |
| B7-H3 | CD47 | HIF1A | KI67 | TIGIT | SDHA |
| BATF3 | CD68 | HLA-DQA1 | LAG3 | Tim3 | UBB |
| BCL2 | CD74 | HLA-DRB1 | LY6E | TNF | ПП 1 |
| CCL5 | CD86 | HLA-E | Multi-CK | VEGFA | ПП 2 |
| CCND1 | CMKLR1 | ICAM1 | NKG7 | НК 1 | ПП 3 |
| CD3E | CSF1R | ICOSLG | pan-Melanoma | НК 2 | ПП 4 |
| CD4 | CTLA4 | IDO1 | PD-1 | НК 3 | ПП 5 |
| CD8A | CTNNB1 | IFNAR1 | PD-L1 | НК 4 | ПП 6 |
| CD11b | CXCL9 | IFNG | PD-L2 | НК 5 | ПП 7 |
| CD11c | CXCL10 | IFNGR1 | PECAM1 | НК 6 | ПП 8 |
| CD20 | CXCR6 | IL6 | PSMB10 | НК 7 | ПП 9 |
| CD27 | DKK2 | IL12B | PTEN | НК 8 | ПП 10 |
3) РНК панели для работы с системами высокопроизводительного секвенирования NGS
| Панель GeoMx Cancer Transcriptome Atlas | Анализ более 1800 генов одновременно с пространственным разрешением в любой области интереса. Всестороннее профилирование опухоли, анализ иммунного статуса и микроокружения |
| Панель GeoMx Host Immune Response Atlas | Анализ более 1800 генов одновременно с пространственным разрешением в любой области интереса. Всестороннее изучение интерферонового сигналинга, активации нейтрофилов, миелоидной активности, анализ генов, связанных специфично с COVID-19 |
| Панель GeoMx Whole Transcriptome Atlas (доступна для заказа с 12.2020) | Полный транскриптом генов на стекле в указанных точках интереса |
Также, компания активно работает в сторону создания набора для пришивания олигонуклеотидов к антителам, что позволит создавать пользовательские белковые панели непосредственно в лаборатории.
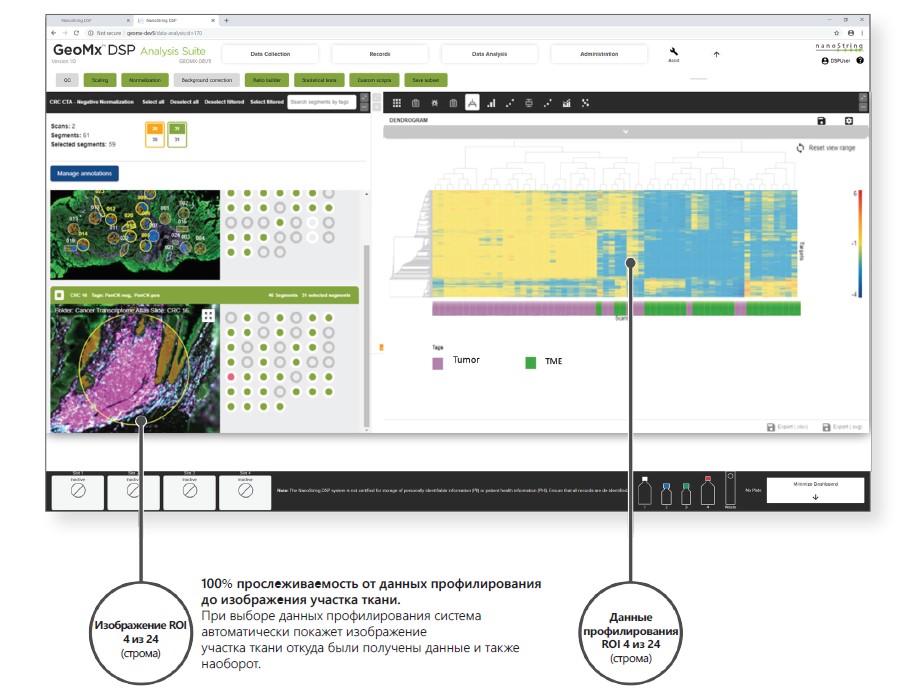
Программное обеспечение GeoMx DSP предоставляет собой комплексное программное решение от сбора изо¬бражений, выбора области интереса до полностью интегрированного анализа данных.
На момент августа 2020 года опубликовано 36 статьей с использованием вышеописанной технологии, не смотря на новизну и официальный анонс только в конце 2019 года. Полный список публикаций https://www.nanostring.com/scientific-content/publications/filterPub/202?filterCategory=59&filterYear=all&filterKeyword=
Современные научные инструменты в генетике и биологии позволяют проводить исследование единичных клеток с высокой точностью. Этот подход является ключевым в медицинской науке, так как метод позволяет выявлять новые мутации, процессы и качества каждой клетки пациентов. Секвенирование генетического материала является центральным проектом в медицинской генетике и онкологии. Эти данные используются для разработки новых методов лечения заболеваний и формирования политики в медицине. Научный подход к исследованию единичных клеток открывает новые возможности в понимании процессов развития и лечения различных заболеваний.
Используемая литература
- «New tools for pathology: a user’s review of a highly multiplexed method for in situ analysis of protein and RNA expression in tissue» Jérémie Decalf , Matthew L. Albert , James Ziai
- Nam JM. Nanoparticle-based bio-bar codes for the ultrasensitive detection of proteins. Science; 2003; 301: 1884–1886. 18. Bao YP, Wei T-F, Lefebvre PA, et al.
- Detection of protein analytes via nanoparticle-based bio bar code technology. Anal Chem 2006; 78: 2055– 2059.
- Wacker R, Ceyhan B, Alhorn P, et al. Magneto Immuno-PCR: a novel immunoassay based on biogenic magnetosome nanoparticles. Biochem Biophys Res Commun 2007; 357: 391–396.
- Fredriksson S, Dixon W, Ji H, et al. Multiplexed protein detection by proximity ligation for cancer biomarker validation. Nat Methods 2007; 4: 327–329.
- Sano T, Smith CL, Cantor CR. Immuno-PCR: very sensitive antigen detection by means of specific antibody-DNA conjugates. Science; 1992; 258: 120– 122.
- Hendrickson ER, Truby TMH, Joerger RD, et al. High sensitivity multianalyte immunoassay using covalent DNA-labeled antibodies and polymerase chain reaction. Nucleic Acids Res 1995; 23: 522–529.
- Agasti SS, Liong M, Peterson VM, et al. Photocleavable DNA barcodeantibody conjugates allow sensitive and multiplexed protein analysis in single cells. J Am Chem Soc 2012; 134: 499–502
- Ullal AV, Peterson V, Agasti SS, et al. Cancer cell profiling by barcoding allows multiplexed protein analysis in fine-needle aspirates. Sci Transl Med 2014; 6: 219ra9.
- Amaria RN, Reddy SM, Tawbi HA, et al. Neoadjuvant immune checkpoint blockade in high-risk resectable melanoma. Nat Med 2018; 24(11): 1649–54.
- Blank CU, Rozeman EA, Fanchi LF, et al. Neoadjuvant versus adjuvant ipilimumab plus nivolumab in macroscopic stage III melanoma. Nat Med 2018; 24: 1655–1661
