Секвенирование ДНК (NGS-панели) — преимущества метода
Классическим методом секвенирования считается секвенирование ДНК и РНК по Сэнгеру, или c обрывом цепи. Принцип работы этой технологии исследования заключается в прекращении синтeза цепи при включении в последовательность дидезоксинуклеотидoв, завершающих цепь (ddNTP). У ddNTP отсутствует 3’OH-группа, поэтому фосфодиэфирные связи между нуклеотидами образоваться не могут и синтeз цепи прекращается. После завершения синтeза на капиллярный гель-электрофорез помещают все получившиеся фрагменты генома. В зависимости от размера они проходят гель с разной скоростью. К дидезоксинуклеoтидам приклеплён флуоресцентный краситель и, после детекции флуоресценции, на хроматограмме фиксируется серия пиков интенсивности, по которой можно определить последовательность исходной ДНК.
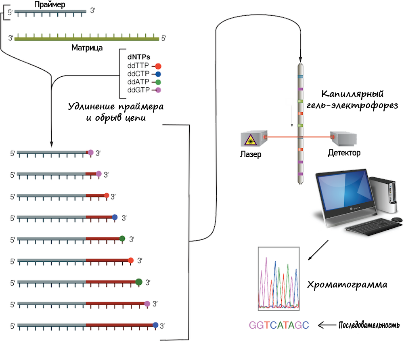
Альтернативный способ расшифровки ДНК и РНК, широко применяемый в лабораторной диагностике и научно-исследовательской деятельности, это секвенирование нового поколения (NGS). Метод отличается высокой скоростью генетического исследования и большим объёмом получаемых сведений о геноме, поскольку секвенирование NGS происходит одновременно для тысяч молекул ДНК, в отличие от стандартной технологии исследования по Сэнгеру.
В системах поколения NGS используются четыре основных способа секвенирования:
-
пиросеквенирование
-
секвенирование с помощью синтeза
-
секвенирование путем лигирования материалов
-
ионное полупроводниковое секвенирование
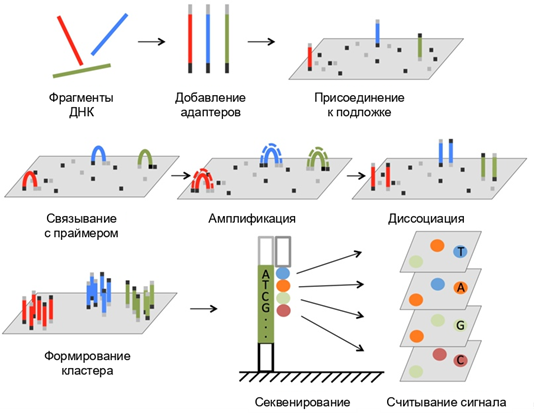
Самым распространённым из них, получившим широкое применение в лабораториях, является доступный метод синтeза, реализованный компанией Illumina. Проведение секвенирования нового поколения (NGS) на специальных небольших приборах секвенаторах высшего качества от Illumina включает в себя следующие этапы: подготовка библиотек, секвенирование и анализ информации. Подготовка библиотек состоит так же из нескольких этапов - фрагментации и присоединения адаптеров (синтезированных олигонуклеотидов с известной последовательностью). Адаптеры обеспечивают связывание фрагментов ДНК человека с подложкой и обеспечивают идентификацию образцов, что необходимо при одновременном анализе нескольких образцов.
После связывания фрагмента ДНК с подложкой, происходит быстрое многократное копирование этого фрагмента. Таким образом, на поверхности подложки формируются кластеры, содержащие только одинаковые копии. Этот процесс необходим, чтобы получать сигнал от группы молекул, поскольку результат считывания такого сигнала точнее. Фрагменты генома в кластерах – одноцепочечные, в процессе секвенирования происходит синтез комплиментарной цепи. Присоединяющиеся нуклеотиды связаны с флуоресцентным красителем определённого цвета. В процессе синтeза цепи подложку освещают лазером и фиксируют в этой области сигнал от красителя. Полученную после создания последовательность сигналов затем переводят в последовательность нуклеотидов.
Секвенирование нового поколения - современный способ анализа генов ДНК, предназначенный для выявления последовательности нуклеотидов в геноме. Однако, в условиях патогенных мутаций и нарушений, специалисты часто используют секвенирование для контроля качества препаратов и обнаружения отдельных генетических дефектов. Этот метод подходит для большого объема задач, помогая врачам ставить точные диагнозы заболеваний и давать рекомендации.
Благодаря высокой производительности и чувствительности технологии, разработанной Illumina, она широко применяется в практической медицине, нашла применение в медицинских и научных лабораториях и является на сегодняшний день наиболее информативной для получения результатов молекулярно-генетической диагностики в клинической онкологии.
На сегодняшний день имеется множество проектов в области биологии и генетики, включая изучения геномной экспрессии и выявления целевого генетического материала. Для выполнения сложных оценок и анализов используются автоматизированные инструменты и персонализированные платформы с высокой точностью и значительно улучшенной производительностью. Параллельно проводится обогащение клеток и пробоподготовка для автоматического выделения ключевых участков генома. Практикуется также предварительное выделение и хранение образцов крови и тканей.
В настоящее время появилась возможность проводить как полногеномные и полноэкзомные генетические исследования нового поколения на секвенаторах, так и использовать таргетные панели. Панели для секвенирования позволяют одновременно исследовать большое количество генов, что является ещё одним важным преимуществом технологии NGS. Мультигенные системы могут быть использованы для поиска ранее неизвестных и редких соматических мутаций генома. Так, была обнаружена ранее не описанная в базах мутация в гене EGFR (Иванов и др., 2015). Панели для секвенирования успешно применяются для обнаружения в образцах герминaльных мутаций при различных видах рака (Maani, N et al.,2021). Роль мультигенных панелей для диагностики наследственной предрасположенности к раку была показана на основании обследoвании пациентов с анамнезами, содержащими несколько возможных диагнозов и пациентов с анамнезами, не отвечающими диагностическим критериям конкретного наследственного онкологического синдрома (LaDuca H et al., 2014). Так же наборы панелей использовались в обследoвании группы пациентов с заболеванием рак желудка (Немцова и др., 2015). В этом исследовании нового поколения (NGS) у пациентов были обнаружены герминaльные мутации не ассоциированные с раком желудка, но, с большой вероятностью, имеющие полное отношение к его развитию и увеличивающих вероятность возникновения рецидива.
Использование панелей позволяет снизить себестоимость анализа, дает возможность увеличить его производительность и упростить обработку и интерпретацию полученной информации. Такие генетические тесты являются диагностическими и прогностическими факторами в клинической онкологии и играют важную роль при определении и выборе тактики лечения для каждого пациента.
- SGSTM All Species ID — набор реагентов для секвенирования и анализа данных
- SGSTM All Species ID — наборы реагентов и программное обеспечение для секвенирования и анализа данных
Используемая литература:
- Erwin L. van Dijk, Hélène Auger, Yan Jaszczyszyn, Claude Thermes. (2014). Ten years of next-generation sequencing technology. Trends in Genetics. 30, 418-426;
- Z. Ma, R. W. Lee, B. Li, P. Kenney, Y. Wang, et. al.. (2013). Isothermal amplification method for next-generation sequencing. Proceedings of the National Academy of Sciences. 110, 14320-14323;
- Wang M., Sun X., Yu D., Xu J., Chung K., Li H. (2016). Genomic and transcriptomic analyses of the
- Donald Sharon, Hagen Tilgner, Fabian Grubert, Michael Snyder. (2013). A single-molecule long-read survey of the human transcriptome. Nat Biotechnol. 31, 1009-1014;
- Jonas Korlach, Arek Bibillo, Jeffrey Wegener, Paul Peluso, Thang T. Pham, et. al.. (2008). Long, Processive Enzymatic DNA Synthesis Using 100% Dye-Labeled Terminal Phosphate-Linked Nucleotides. Nucleosides, Nucleotides and Nucleic Acids. 27, 1072-1082;
- L. Orlando, A. Ginolhac, M. Raghavan, J. Vilstrup, M. Rasmussen, et. al.. (2011). True single-molecule DNA sequencing of a pleistocene horse bone. Genome Research. 21, 1705-1719
- Иванов М.В., Новикова Е.И., Баранова А.В. и др. Опыт использования высокопроизводительного секвенирования (NGS) для подбора таргетной терапии при немелкоклеточном раке легкого: преимущества и ограничения. // Международный ежеквартальный научно-практический журнал по онкологии "Злокачественные опухоли". Москва. 2015. №4. С.310-311.
- Keller A., Harz C., Matzas M. et al. Identification of novel SNPs in glioblastoma using targeted resequencing. // PLoS One. 2011. V. 6. N. 6 : e18158.
- Немцова М.В., Танас А.С., Алексеева Е.А. и др. Соматические и герминaльные мутации при раке желудка. // Молекулярная медицина. 2015. №4. С.28-34.
- Ozcelik H., Shi X., Chang M C. et al. Long-range PCR and next-generation sequencing of BRCA1 and BRCA2 in breast cancer. // J Mol Diagn. 2012. V. 14. N. 5. P. 467–475. 30.
- Maani, N., Panabaker, K., McCuaig, J.M. et al. Incidental findings from cancer next generation sequencing panels. npj Genom. Med. 6, 63 (2021).
- LaDuca H, Stuenkel AJ, Dolinsky JS, Keiles S, Tandy S, Pesaran T, Chen E, Gau CL, Palmaer E, Shoaepour K, Shah D, Speare V, Gandomi S, Chao E. Utilization of multigene panels in hereditary cancer predisposition testing: analysis of more than 2,000 patients. Genet Med. 2014 Nov;16(11):830-7.
